选取了不同类型、地域来源的68份的野生、111份栽培(包括农家种、现代品种及青稞)材料的共179份大麦为研究对象,开展了重测序分析(图 1),共鉴定到约5,400万个SNP位点,系统进化树、主成分及ADMIXTURE结果可将所有材料依据不同类型、居群及地理来源划分,驯化过程中栽培大麦丢失了约21.97%的遗传多样性。全基因组选择性消除分析鉴定到1,672个驯化和896个改良相关候选基因。进一步对株高、穗长、千粒重、粒长宽比等农艺及籽粒性状,以及盐胁迫下光合效率、叶片保护酶、丙二醛含量、过氧化氢酶、超氧化物歧化酶等生理生化指标,共计27个性状进行了全基因组关联分析,鉴定得到与大麦重要性状相关遗传变异位点和候选基因。该研究在全基因组水平系统揭示了大麦驯化的分子基础,也为大麦分子改良及育种提供候选参考。
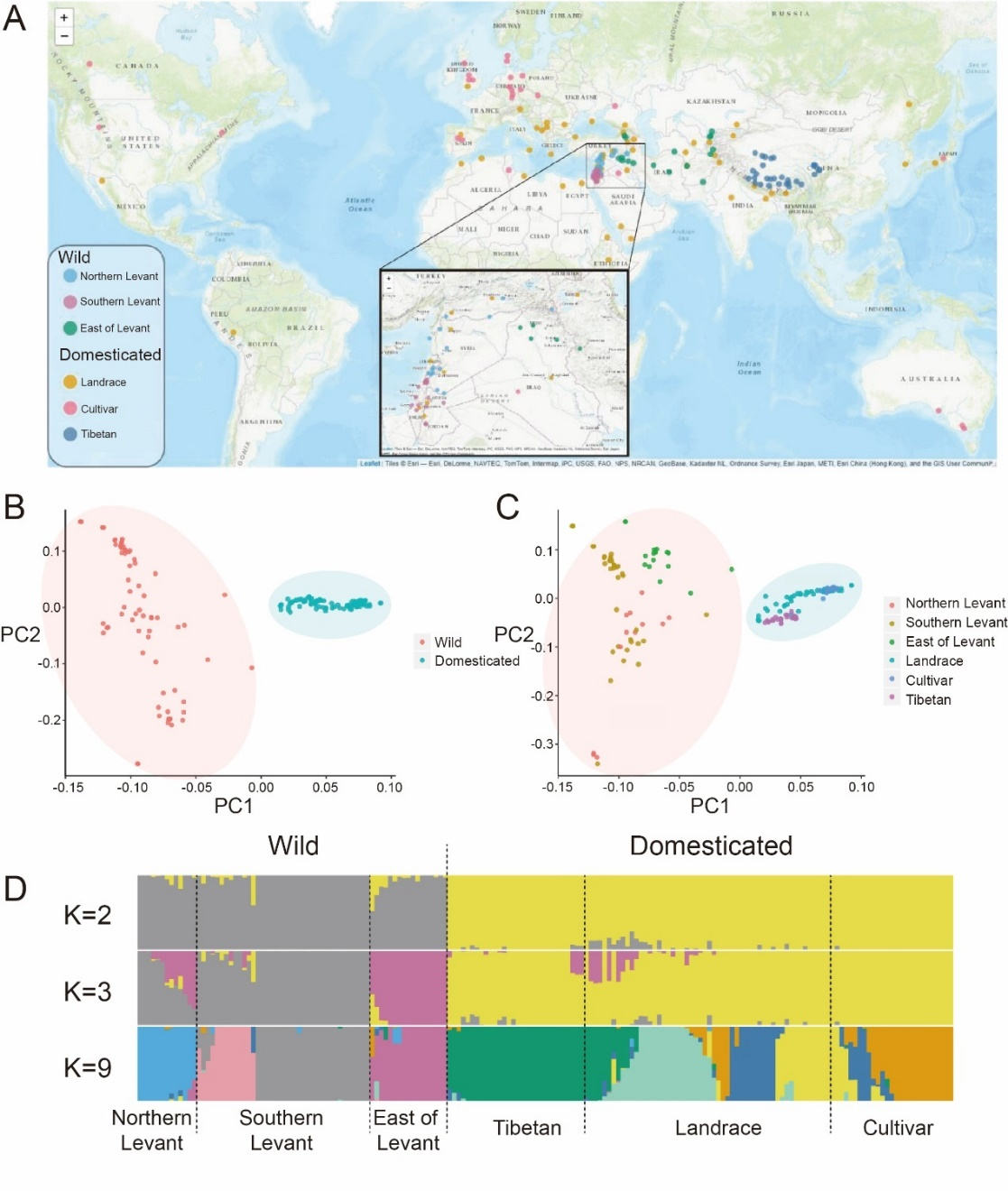
图 1. 179份大麦材料的系统进化及群体遗传结构分析
课题组及其合作者基于上述研究,筛选多态性好、代表性强、覆盖均匀的40,519个SNP位点,利用GenoBaits技术开发了大麦40K SNP液相芯片,对380份不同大麦材料的测试表明,该芯片在基因分型、种群结构分析等方面均有良好的表现(图 2)。GWAS鉴定到一系列与重要性状相关的选择信号区间和基因,例如,我们鉴定到132个和187个穗数和穗长相关的信号,其中包括一些已知的与穗数相关候选基因,如HvPRF5、HvYAO、HvCYP71B7、HvYUC2、HvSEC3A及HvBB等,进一步构建了大麦的高密度遗传图谱。大麦40K液相芯片的开发填补了我国大麦育种领域相关工具的空缺,为打破大麦育种瓶颈、提高育种效率、实现大麦育种突破提供了重要工具。

图2. 大麦40K SNP液相芯片的开发及农艺性状的全基因组关联分析

